Gromacs系列
该镜像集成开源高性能分子动力学模拟软件,通过强化多精度算法与GPU并行加速(支持NVIDIA/AMD显卡)实现纳秒级生物大分子运动模拟,优化AI辅助建模与超大体系计算效率,提供跨平台开箱即用解决方案
 10
100元/小时
v2025.4
v2024.2
v2021.03
v1.0
Gromacs系列
镜像简介
本镜像是专为GROMACS分子动力学模拟打造的优化环境,提供高性能、灵活且开源的仿真计算能力。支持生物大分子、材料体系等多尺度模拟,集成GPU加速与常用分析工具,适用于生物医药、化学材料及物理科研领域的研究与教学,助力用户高效完成复杂的分子模拟计算任务。
镜像使用指南
创建实例
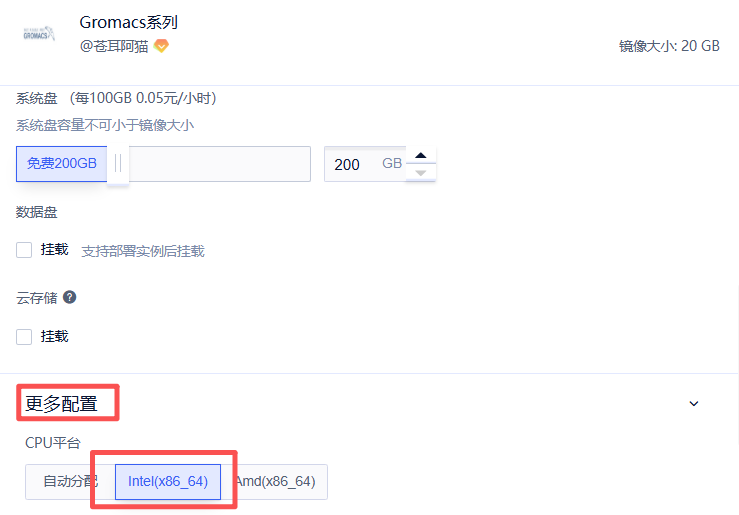
选择RTX40系列,点击下方更多配置,选择Intel机型,一定要选择intel机型,否则无法正常运行!
准备工作
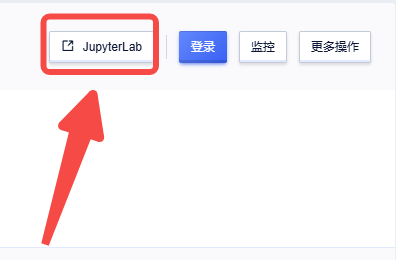
点击【JupyterLab】,创建一个新目录:
mkdir /workspace/test_run
cd /workspace/test_run
第一步:获取结构文件 (PDB)
官方示例使用 1AKI (溶菌酶)。
wget https://files.rcsb.org/download/1AKI.pdb
第二步:生成拓扑 (pdb2gmx)
这一步将 PDB 转换为 GROMACS 能识别的拓扑文件 (topol.top) 和坐标文件。
- 输入
15选择 OPLS-AA/L 力场。 -water spce选择 SPCE 水模型。
gmx_mpi pdb2gmx -f 1AKI.pdb -o 1AKI_processed.gro -water spce <<EOF
15
EOF
第三步:定义模拟盒子 (editconf)
定义一个盒子,让蛋白质居中,并且边缘距离蛋白质至少 1.0 nm。
gmx_mpi editconf -f 1AKI_processed.gro -o 1AKI_newbox.gro -c -d 1.0 -bt cubic
第四步:填充溶剂 (solvate)
向盒子中加入水分子。
gmx_mpi solvate -cp 1AKI_newbox.gro -cs spc216.gro -o 1AKI_solv.gro -p topol.top
第五步:添加离子 (genion)
此时系统带电,必须加入离子使其电中性。
我们需要先创建一个简单的 .mdp 文件来生成离子的运行文件。
-
创建 ions.mdp:
cat > ions.mdp <<EOF ; 离子添加参数 integrator = steep emtol = 1000.0 emstep = 0.01 nsteps = 50000 nstlist = 1 cutoff-scheme = Verlet ns_type = grid coulombtype = cutoff rcoulomb = 1.0 rvdw = 1.0 pbc = xyz EOF -
生成 TPR 文件:
gmx_mpi grompp -f ions.mdp -c 1AKI_solv.gro -p topol.top -o ions.tpr -maxwarn 2 -
替换水分子为离子:
- 输入
13(SOL) 表示将水分子替换为离子。 -neutral选项会自动计算需要多少正/负离子来平衡电荷。
gmx_mpi genion -s ions.tpr -o 1AKI_solv_ions.gro -p topol.top -pname NA -nname CL -neutral <<EOF 13 EOF - 输入
第六步:能量最小化 (Minimization)
在正式跑模拟之前,必须消除不合理的原子重叠。
-
创建 minim.mdp (复用 ions.mdp 即可,内容基本一样,或者重新写一个标准的):
cat > minim.mdp <<EOF integrator = steep emtol = 1000.0 emstep = 0.01 nsteps = 50000 nstlist = 1 cutoff-scheme = Verlet rlist = 1.0 coulombtype = PME rcoulomb = 1.0 rvdw = 1.0 pbc = xyz EOF -
预处理:
gmx_mpi grompp -f minim.mdp -c 1AKI_solv_ions.gro -p topol.top -o em.tpr -
运行最小化: 这一步很快,主要是为了让系统稳定。
gmx_mpi mdrun -v -deffnm em
第七步:正式运行 (Minimal MD)
官方流程通常还要经过 NVT/NPT 平衡,但作为最小示例,我们可以直接跑一个简短的动力学模拟。
-
创建 md.mdp:
cat > md.mdp <<EOF integrator = md dt = 0.002 nsteps = 50000 ; 100 ps nstxout = 5000 nstvout = 5000 nstfout = 5000 nstenergy = 5000 nstlog = 5000 cutoff-scheme = Verlet nstlist = 20 rlist = 1.0 coulombtype = PME rcoulomb = 1.0 rvdw = 1.0 tcoupl = V-rescale tc-grps = Protein Non-Protein tau_t = 0.1 0.1 ref_t = 300 300 pbc = xyz dispcorr = EnerPres EOF -
预处理:
gmx_mpi grompp -f md.mdp -c em.gro -p topol.top -o md.tpr -maxwarn 2 -
在 4090 上全速运行:
gmx_mpi mdrun -deffnm md -nb gpu -pme gpu -update gpu -bonded gpu
总结
这就是 GROMACS 官方文档中标准的从 PDB 到 MD 的全流程。
@苍耳阿猫 认证作者
认证作者
 认证作者
认证作者
镜像信息
已使用108 次
运行时长
2003 H
镜像大小
20GB
最后更新时间
2026-01-27
支持卡型
RTX40系
+1
框架版本
CUDA版本
12.8
应用
JupyterLab: 8888
版本
v2025.4
2026-01-27
v2024.2
2025-12-18
v2021.03
2026-02-02
查看全部
